Model jazyka proteinů společnosti EvolutionaryScale – jeden z největších modelů umělé inteligence v biologii – vytvořil nové fluorescenční proteiny a získal velké investice.
Autor: Ewen Callaway
příroda
Model [umělé inteligence](https://www.nature.com/subjects/machine-learning) (AI), který hovoří [jazykem proteinů](https://www.nature.com/subjects/protein-design) — jeden z největších, které byly dosud vyvinuty pro biologii — byl použit k vytvoření nových fluorescenčních molekul.
Tento měsíc to oznámila společnost EvolutionaryScale v New Yorku spolu s novými finančními prostředky ve výši 142 milionů dolarů, které budou použity na [vývoj léků](https://www.nature.com/articles/d41586-023-02227-y), udržitelnost a další aktivity. Společnost, kterou založili vědci, kteří dříve pracovali v technologickém gigantu Meta, je nejnovějším účastníkem na stále přeplněnějším poli, kde se na biologická data aplikují špičkové modely strojového učení vycvičené na jazyce a obrazech.
„Chceme vytvořit nástroje, které umožní programovat biologii,“ říká Alex Rives, hlavní vědecký pracovník společnosti, který se podílel na úsilí společnosti Meta o aplikaci umělé inteligence na biologická data.
Nástroj umělé inteligence společnosti EvolutionaryScale, nazvaný ESM3, je tzv. proteinový jazykový model. Byl vycvičen na více než 2,7 miliardách proteinových sekvencí a struktur a také na informacích o funkcích těchto proteinů. Model lze použít k [vytváření proteinů](https://www.nature.com/articles/d41586-023-02227-y) podle specifikací zadaných uživateli, podobně jako text vyplivnutý chatboty, jako je ChatGPT.
„Bude to jeden z modelů umělé inteligence v biologii, kterému budou všichni věnovat pozornost,“ říká Anthony Gitter, počítačový biolog z University of Wisconsin–Madison.
Zářící nahoru
Rives a jeho kolegové pracovali na dřívějších verzích modelu ESM ve společnosti Meta, ale v loňském roce se vydali na vlastní pěst poté, co Meta ukončila svou práci v této oblasti. Již dříve použili model ESM-2 k vytvoření [volně dostupné databáze 600 milionů předpovězených proteinových struktur](https://www.nature.com/articles/d41586-022-03539-1)[1](https://www.nature.com/articles/d41586-024-02214-x?utm_source=tldrnewsletter#ref-CR1). Jiné týmy od té doby použily verze modelu ESM-1 k [návrhu protilátek s lepší aktivitou proti patogenům](https://www.nature.com/articles/d41586-023-01516-w) včetně SARS-CoV-2[2](https://www.nature.com/articles/d41586-024-02214-x?utm_source=tldrnewsletter#ref-CR2) a k přepracování proteinů „anti-CRISPR“ s cílem zlepšit účinnost nástrojů pro úpravu genů[3](https://www.nature.com/articles/d41586-024-02214-x?utm_source=tldrnewsletter#ref-CR3).
V letošním roce použila další společnost zabývající se umělou inteligencí v oblasti biologie, Profluent v kalifornském Berkeley, svůj vlastní model proteinového jazyka k [vytvoření nových proteinů pro úpravu genů inspirovaných CRISPR](https://www.nature.com/articles/d41586-024-01243-w) a jednu takovou molekulu poskytla k volnému použití.
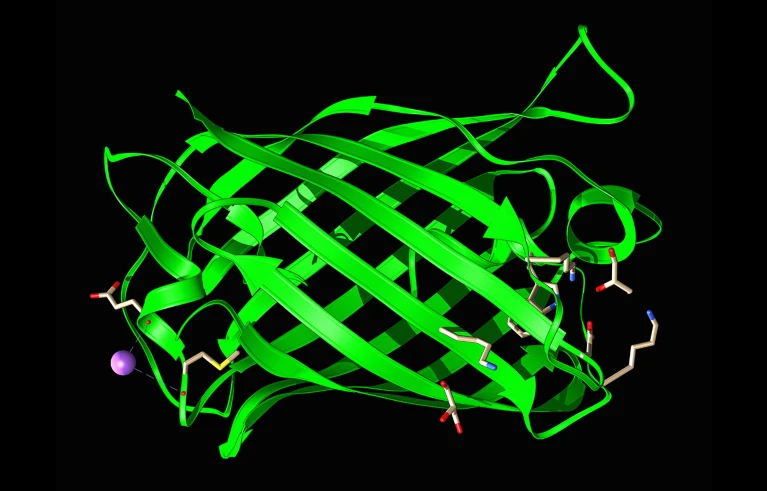
Aby demonstroval svůj nejnovější model, rozhodl se Rivesův tým přepracovat dalšího biotechnologického koně: zelený fluorescenční protein (GFP), který absorbuje modré světlo a svítí zeleně. Vědci izolovali GFP v 60. letech 20. století z bioluminiscenční medúzy _Aequorea victoria_. Pozdější práce – která byla za tento objev oceněna Nobelovou cenou – ukázala, jak může GFP označovat jiné proteiny pozorované pod mikroskopem, vysvětlila molekulární podstatu jeho fluorescence a vyvinula syntetické verze proteinu, které svítí mnohem jasněji a v různých barvách.
Vědci od té doby identifikovali další podobně tvarované fluorescenční proteiny, které mají společné jádro „chromoforu“ pohlcujícího a vyzařujícího světlo a obklopeného soudkovitým lešením. Rivesův tým požádal ESM3 o vytvoření příkladů proteinů podobných GFP, které by obsahovaly sadu klíčových aminokyselin, jež se nacházejí v chromoforu GFP.
Výzkumníci syntetizovali 88 nejslibnějších návrhů a měřili jejich schopnost fluoreskovat. Většina z nich byla falešná, ale jeden návrh, nepodobný známým fluorescenčním proteinům, svítil slabě – asi 50krát slaběji než přirozené formy GFP. S využitím sekvence této molekuly jako výchozího bodu výzkumníci pověřili ESM3, aby vylepšil svou práci. Když vědci vytvořili asi 100 výsledných návrhů, několik z nich bylo stejně jasných jako přírodní GFP, které jsou však stále výrazně slabší než laboratorně vytvořené varianty.
Jeden z nejjasnějších proteinů navržených ESM3, nazvaný esmGFP, má podle předpokladů strukturu podobnou těm přírodním fluorescenčním proteinům. Jeho aminokyselinová sekvence je však značně odlišná a odpovídá méně než 60 % sekvence nejpříbuznějšího fluorescenčního proteinu v souboru trénovacích dat. V preprintu zveřejněném na serveru bioRxiv[4](https://www.nature.com/articles/d41586-024-02214-x?utm_source=tldrnewsletter#ref-CR4) Rives a jeho kolegové uvádějí, že na základě rychlosti přirozených mutací odpovídá tato úroveň sekvenčního rozdílu „více než 500 milionům let evoluce“.
Gitter se však obává, že toto srovnání je neužitečný a potenciálně zavádějící způsob popisu produktu špičkového modelu umělé inteligence. „Zní to děsivě, když se zamyslíte nad UI a zrychlující se evolucí,“ říká. „Mám pocit, že přehnané popisování toho, co model umí, může oboru uškodit a může být nebezpečné pro veřejnost.“
Rives vidí generování nových proteinů pomocí iterací různých sekvencí v modelu ESM3 jako obdobu evoluce. „Myslíme si, že perspektiva toho, co by příroda potřebovala, aby něco takového vytvořila, je zajímavá,“ dodává.
Práh rizika
ESM3 je jedním z prvních biologických modelů umělé inteligence, který při svém tréninku využívá dostatečný výpočetní výkon, aby vývojáři museli na základě prezidentského nařízení z roku 2023 informovat vládu USA a nahlásit [opatření ke zmírnění rizik](https://www.nature.com/articles/d41586-024-00699-0). Společnost EvolutionaryScale uvádí, že již byla v kontaktu s americkým Úřadem pro vědeckou a technologickou politiku.
Verze ESM3, která tuto hranici překonala – obsahuje téměř 100 miliard parametrů neboli proměnných, které model používá k vyjádření vztahů mezi sekvencemi -, není veřejně dostupná. U menší verze s otevřeným zdrojovým kódem byly některé sekvence, například sekvence virů a seznam znepokojivých patogenů a toxinů sestavený vládou USA, z tréninku vyloučeny. Ani ESM3-open – který si mohou vědci kdekoli stáhnout a spustit nezávisle – nemůže být vyzván ke generování takových proteinů.
Martin Pacesa, strukturní biolog ze Švýcarského federálního technologického institutu v Lausanne, je nadšený, že může začít pracovat s ESM3. Je to jeden z prvních biologických modelů, který vědcům umožňuje specifikovat návrhy pomocí popisu jejich vlastností a funkcí v přirozeném jazyce, poznamenává, a je zvědavý, jak se tato a další funkce osvědčí experimentálně.
Pacesa je ohromen tím, že společnost EvolutionaryScale zveřejnila open-source verzi ESM3 a jasný popis toho, jak byla tato největší verze vyškolena. Říká však, že samostatný vývoj největšího modelu by si vyžádal obrovské výpočetní prostředky. „Žádná akademická laboratoř ho nebude schopna zopakovat.“
Rives by rád použil ESM3 i na další návrhy. Pacesa, který byl členem týmu, jenž použil jiný model proteinového jazyka pro tvorbu nových proteinů CRISPR, říká, že bude zajímavé sledovat, jak si ESM3 povede v této oblasti. Rives předpokládá využití v oblasti udržitelnosti – video na webových stránkách společnosti ukazuje návrh enzymů požírajících plasty – a při vývoji protilátek a dalších léků na bázi proteinů. „Je to skutečně model na hranici,“ říká.

I don’t think the title of your article matches the content lol. Just kidding, mainly because I had some doubts after reading the article.